利用数据库数据进行定制化的生信分析
让您的文字更出彩
|
DNA甲基化(DNA methylation)DNA甲基化(DNA methylation)是基因调控的手段之一,在维持细胞正常功能、传递基因组印记、胚胎发育、肿瘤发生等方面起着至关重要的作用,更是表观遗传学研究的热点。全基因组甲基化测序(Whole Genome Bisulfite Sequencing,WGBS)作为甲基化测序的“金标准”,对有参考基因组的物种在全基因组水平进行全面、高效、高准确度的甲基化研究,从而构建单碱基分辨率的全基因组DNA甲基化水平图谱。 哺乳动物DNA甲基化主要为CG型,而富含CpG二核苷酸的CpG岛常位于转录调控区附近,与56%的人类基因组编码基因相关。Agilent公司推出的CpG岛精准甲基化测序,采用SureSelect系统进行靶标富集,全面覆盖基因组中最具表观遗传学研究意义的区域,极大提高测序数据有效性,达到单碱基分辨率,同时降低成本,缩短测序时长,实现多样本实验。
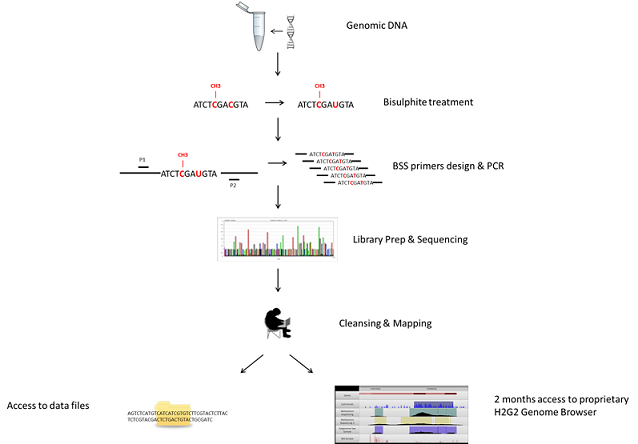
A workflow for Bisulfite Sequencing 我们的优势1. 精准覆盖:采用SureSelect靶标富集系统,可覆盖370万CpG,包括CpG岛、GENCODE启动子、癌症以及组织特异性DMR等,对具有生物学意义的甲基化位点实现全面覆盖; 2. 高深度:只需10Gb数据可达100×覆盖深度,是普通测序深度的3倍,极大提高了对低频甲基化位点的探测率; 3. 高灵敏度:采用Bisulfite转化前捕获,对甲基化和非甲基化均等捕获;单碱基分辨率,可精准检测每个胞嘧啶的甲基化状态; 4. 低成本、高效率:使用液体捕获技术,大大提高捕获效率;同时靶标区域的选择策略可降低成本,得以实现多样本快速实验; 样本要求组织样品≥50mg DNA样品 1. 请提供总量≥3μg/样本,浓度≥100ng/μL的DNA; 2. OD260/280介于1.8-2.0之间; 3. 电泳检测无明显RNA污染,基因组条带清晰、完整,无降解; 4. 送样时请标记清楚样品编号,管口使用Parafilm膜密封; 5. 样品保存期间切忌反复冻融; 6. 送样时请使用干冰运输。 注:不同样品之间存在差异,详情请向我们咨询 实验流程
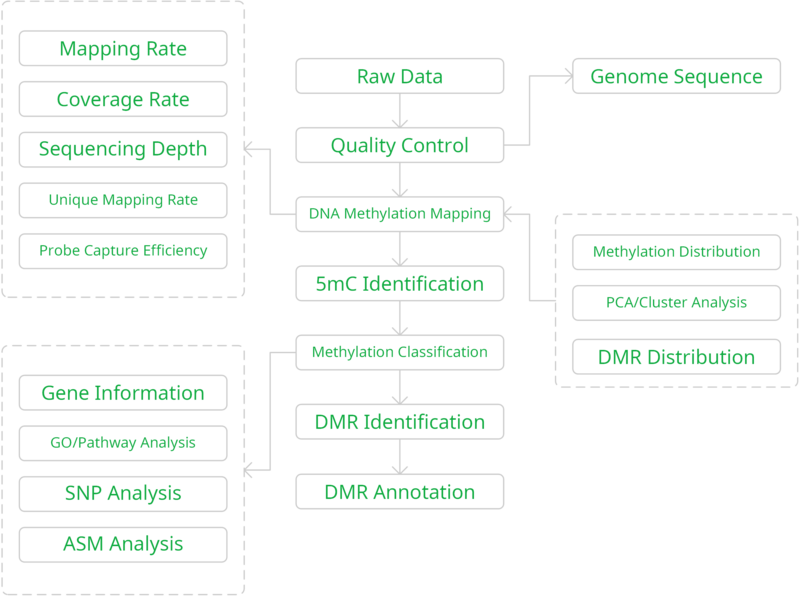
1. DNA提取及质控:凝胶电泳质控→Nanodrop质控→Agilent 2200质控; 2. DNA超声片段化:片段化后,末端修复加A,并连接甲基化接头; 3. SureSelect靶向捕获:单链RNA探针杂交进行液相捕获; 4. Bisulfite转化:靶向捕获之后进行BS转化,降低甲基化偏好性和DNA损伤; 5. 加接头扩增:捕获之后进行PCR扩增文库构建,降低PCR偏好性; 6. 上机测序:我们建议选择NovaSeq,双端测序,通量大,碱基精度高,而且成本低,速度快。数据量:10G。 数据分析流程
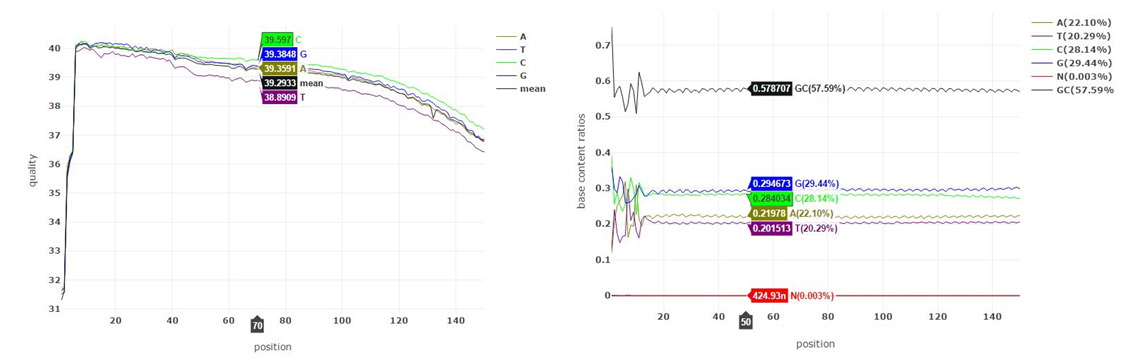
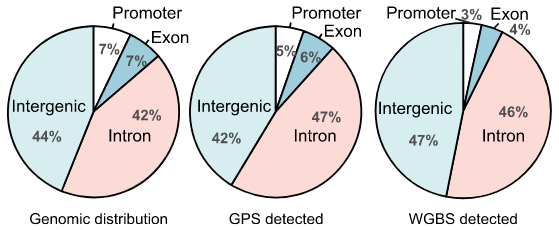
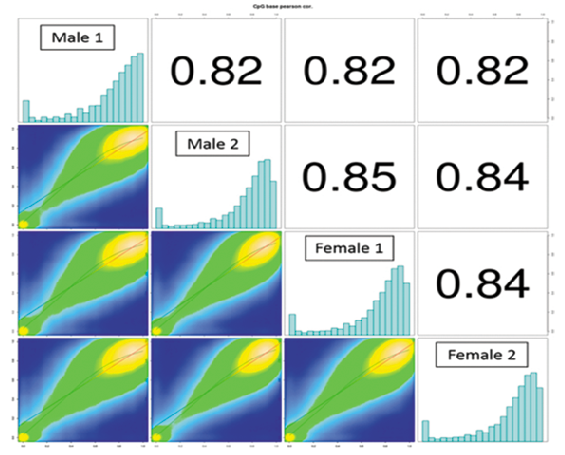
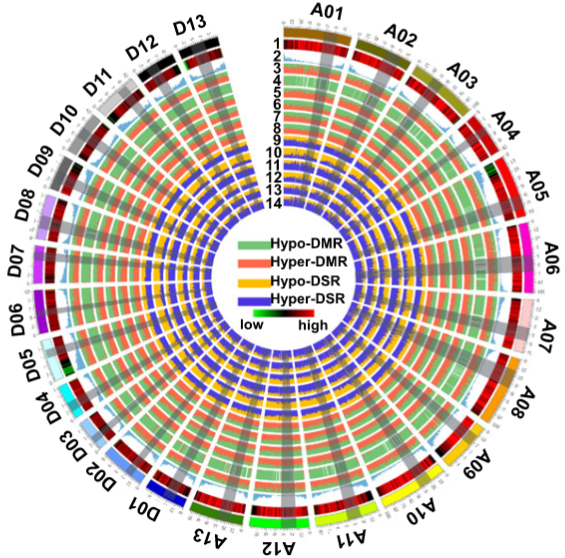
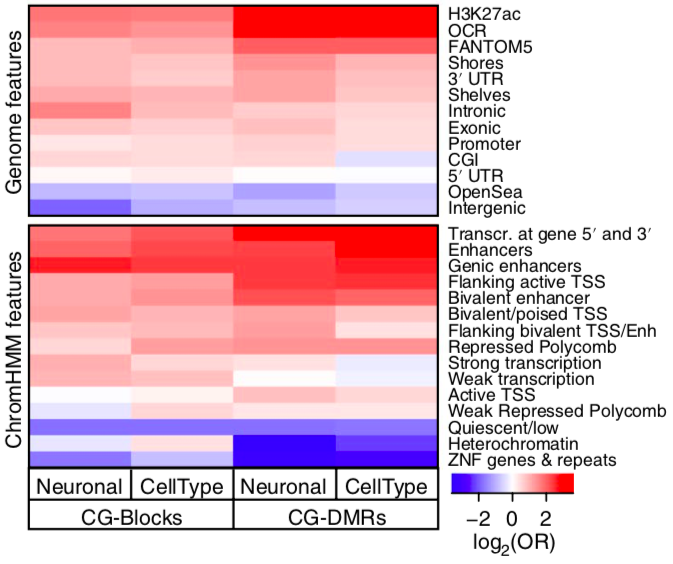
结果示例1、原始数据质量控制针对过短序列,污染序列,尾端低质量序列,进行下机原始数据(Raw Data)的过滤,并采用Fastp对于过滤前后的数据进行质控,得到碱基质量,GC含量,长度分布等分析结果。图1部分展示Raw Data的质量控制结果。 碱基质量结果图 注:左图横坐标代表碱基位点,纵坐标代表碱基质量值,不同颜色曲线代表不同碱基在每条read上的质量值;右图横坐标代表碱基位点,纵坐标代表碱基含量比值,不同颜色曲线代表不同位点各碱基含量。 2、序列比对(DNA Mapping)采用Bismark等软件将过滤后的数据链特异性地比对到参考基因组上,得到5mC的甲基化类型、状态、比例,用于后续甲基化水平、分布及差异甲基化分析。采用Samtools工具,对于基因组比对文件,进行排序并建立索引。 比对流程 注:该图为Bismark比对原理图 3、甲基化水平及分析采用Bismark算法对5mC鉴定分析结果进行甲基化类型、水平和分布的分析,得到在不同样本中的甲基化类型、水平及其基因组上的分布情况。 CpG位点在基因组上的分布 Li J et al., Genome Research. 2019 注:该图对比了GPS和WGBS检测出的CpG位点在基因组上的分布情况 4、样本相关性、聚类、PCA分析以每一个样本的5mC鉴定分析结果为研究对象,采用PCA降维算法,Pearson相关性系数,K-Mean聚类等多种算法,对于每一个甲基化测序样本的甲基化程度以及样本相关性进行描述,得到样本的关联结果。 多样本间甲基化相关性分析 Wan et al., Sci Rep. 2016 注:该图展示了罗非鱼骨骼肌雌雄两性间CpG甲基化的Pearson相关性 5、差异甲基化区域(DMR)筛选以每一个样本的5mC鉴定分析结果为研究对象,采用Methykit算法,对于差异甲基化区域进行分析,得到不同样本间差异的甲基化区域。 CIRCOS:差异甲基化区域 Yizan Ma et al., Plant Cell. 2018 注:通过绘制可视化Circos图,展示了常温(NT)和高温(HT)处理对不同品系棉花(84021和H05)花药不同时期DMR在染色体上的分布情况 6、DMS/DMR相关基因分析依据DMR注释获取DMR相关的基因群,然后针对该基因群进行相关基因群的GO、KEGG Pathway和PPI分析。 DMR相关基因群PPI分析 注:基于DMS/DMR注释基因的GO条目和Pathway条目,我们可以根据其上下级调控关系绘制GO-Tree和Pathway-Act-Network,并基于STRING数据库进行PPI分析 1、差异甲基化区域(DMR)注释采用ChipSeeker算法,对于差异甲基化区域进行注释分析,得到DMR对应的基因以及所在的基因结构,包括启动子、外显子、内含子、基因间区等。 不同样本CG-DMR的基因组分布 Rizzardi LF et al., Nat Neurosci. 2019 注:该图通过聚类分析展示了不同Cell type和neuronal中的CG-DMR在基因组结构上的分布情况 文献示例[1] Ai T, et al. DNA methylation profile is associated with the osteogenic potential of three distinct human odontogenic stem cells. Signal Transduct Target Ther. 2018. Jan 12;3:1. [2] Liu H, et al. Integrative analysis of DNA methylation, mRNAs, and small RNAs during maize embryo dedifferentiation. BMC Plant Biol. 2017 Jun 15;17(1):105. [3] Fang X, et al. Comparative genome-wide methylation analysis of longissimus dorsi muscles between Japanese black (Wagyu) and Chinese Red Steppes cattle. PloS One. 2017 Aug 3;12(8):e0182492. [4] Wang Z, et al. Decreased Methylation Level of H3K27me3 Increases Seizure Susceptibility. Mol Neurobiol. 2017 Nov;54(9):7343-7352. [5] Cheng J, et al. SUMOylation of MeCP2 is essential for transcriptional repression and hippocampal synapse development. J Neurochem. 2014 Mar;128(6):798-806. |