利用数据库数据进行定制化的生信分析
让您的文字更出彩
|
单细胞测序技术(single cell sequencing)单细胞测序技术(single cell sequencing)是指在单个细胞水平上,对基因组、转录组、表观组进行高通量测序分析的一项新技术,它能够弥补传统高通量测序的局限性,揭示单个细胞的基因结构和基因表达状态,反映细胞间的异质性。2013年,单细胞测序技术被《Science》将其列为年度最值得关注的六大领域榜首;2015年再次登上Science转化医学封面。目前,单细胞测序技术在肿瘤、发育生物学、微生物学、神经科学、以及植物学等领域发挥重要作用,正成为生命科学研究的焦点,具有广阔的应用前景。
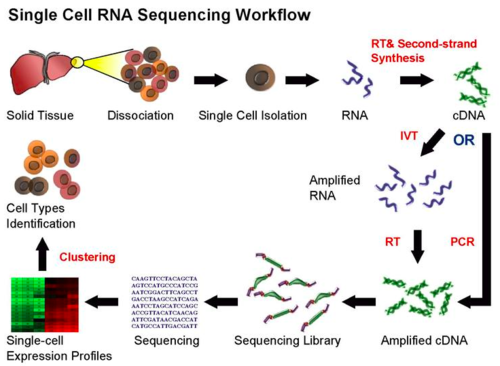
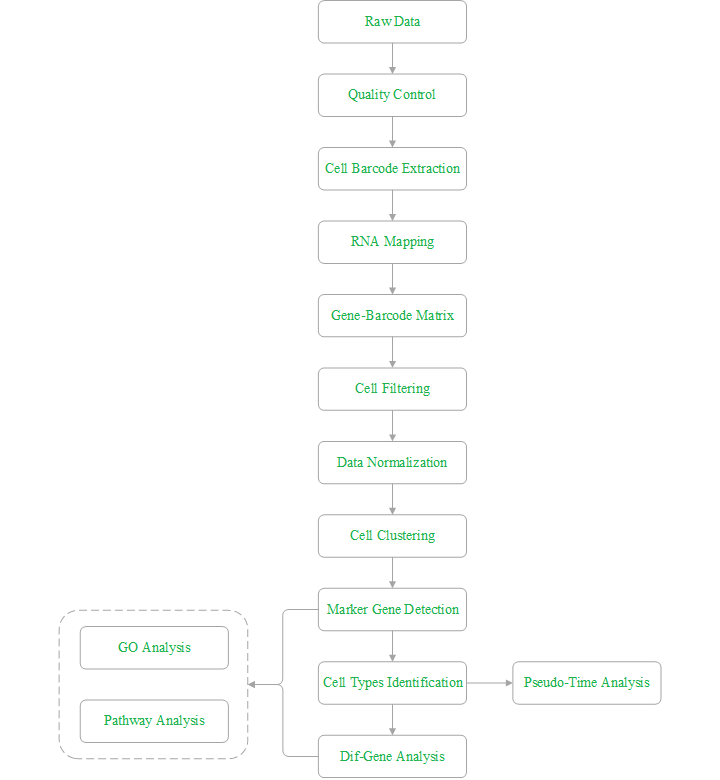
单细胞转录组测序流程--点我了解更多 我们的优势1. 拥有行业认可的10X Genomics和BD Rhapsody单细胞平台,实现真正意义上的单细胞测序; 2. 具有丰富的、针对不同样本类型的单细胞悬液制备经验,如:外周血、细胞系、新鲜/冻存器官组织、肿瘤组织,确保细胞活性达到测序要求; 3. 拥有成熟的单细胞测序文库构建技术,一次性完成1000-10000个细胞的文库构建,真正测全组织中所有细胞类型,做到对样本中所有类型细胞的全面解析; 4. 具备完善的单细胞测序和实验质控流程,以及丰富的单细胞测序和数据分析经验,实现个性化、定制化数据分析服务。 样本要求样本类型: 组织、血液、培养的细胞系、制备好的单细胞悬液 注:若客户样本为组织,且无能力进行组织解离来获取单细胞悬液,烈冰将尽可能提供技术及实验上的帮助,但因不同类型样本的特异性,无法保证实验方法适用于所有类型组织。 质量要求: 1. 细胞活性大于70% 2. 浓度为500-2000细胞/μl 3. 体积不小于200μl 4. 细胞培养基及缓冲液不能含Ca2+和Mg2+ 5. 细胞体积小于40μm 实验流程1. 单细胞悬液制备:根据样本特性选择合适的单细胞悬液制备方法,注意红细胞裂解;如若客户样本为已经制备好单细胞悬液,该步骤可以省略; 2. 细胞活性检测:细胞活性需大于70%; 3. 单细胞捕获:通过分选平台(BD、10X、Drop-seq)对每个细胞进行捕获; 4. 细胞/转录本标签添加:对结合磁珠标签的RNA进行逆转录引入CB和UMI; 5. 文库构建:对cDNA进行随机引物PCR扩增; 6. 上机测序:烈冰推荐Illumina Hiseq或NovaSeq测序平台,数据量100G/样本。 数据分析流程
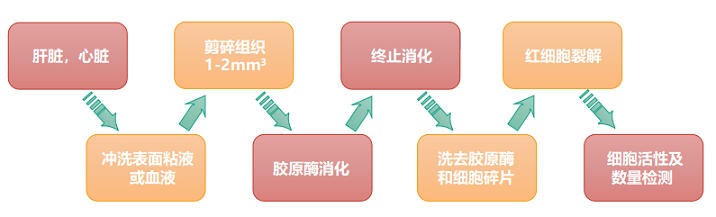
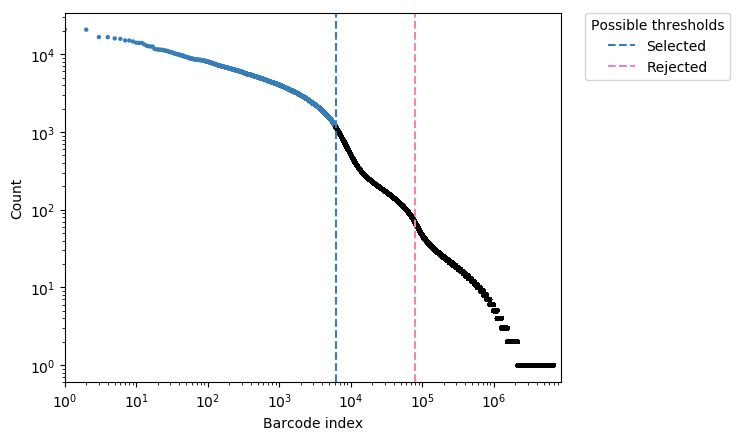
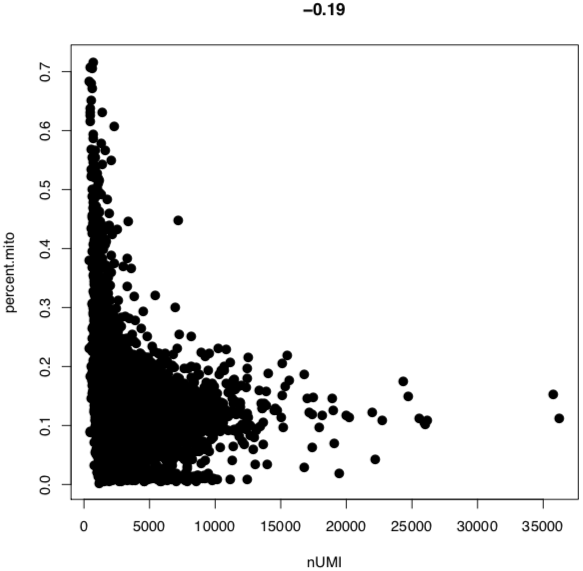
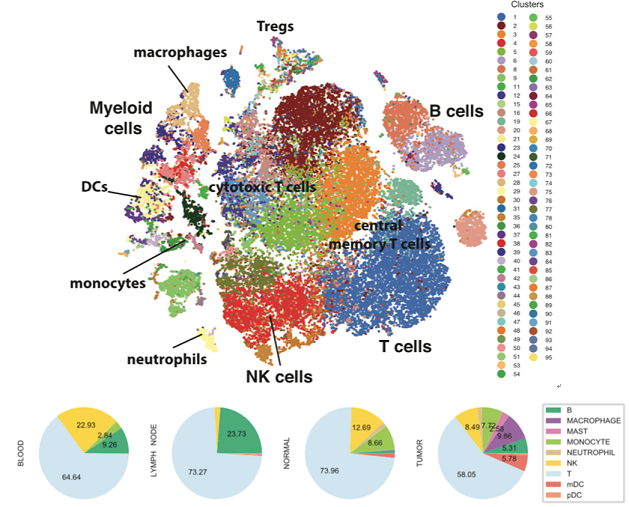
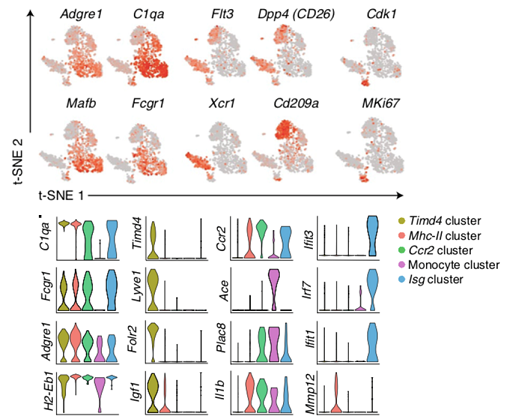
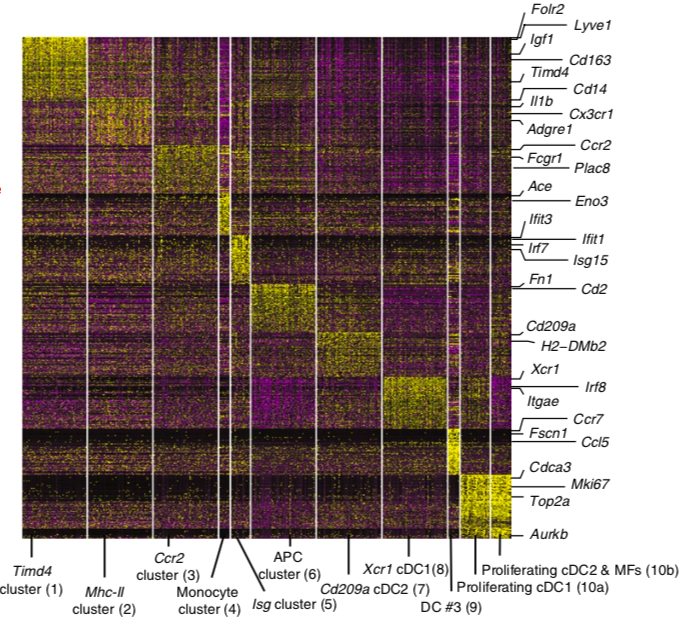
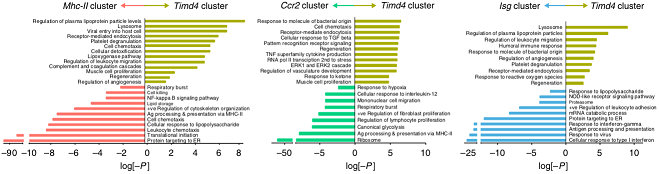
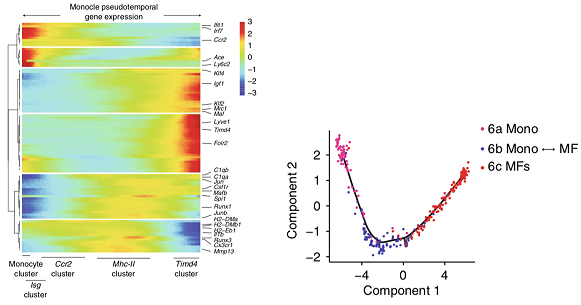
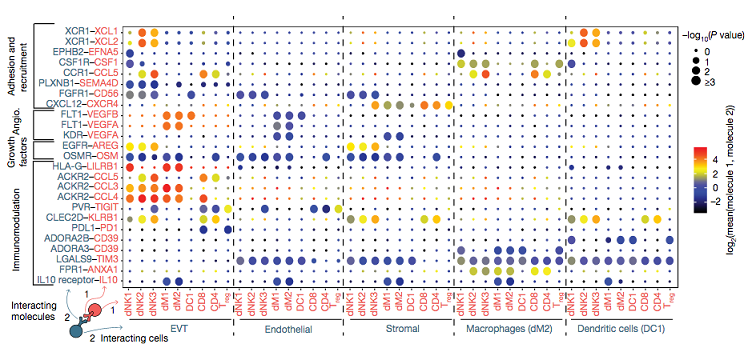
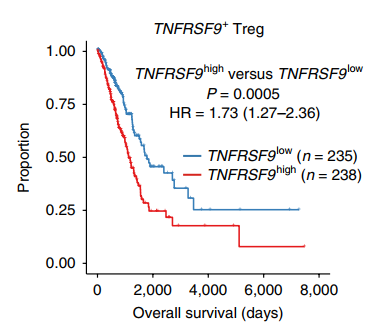
结果示例1、单细胞悬液制备(附加收费项目)根据研究目的和对象获取新鲜的样本,包括不同类型的肿瘤样本及其对应的正常样本。再根据组织样本的特性,选择合适的消化条件(酶液、消化温度和时间),过筛后获得所需单细胞悬液。 单细胞悬液的制备流程 2、单细胞计数和活性检测将细胞从培养液体系更换为上样缓冲液体系(Sample Buffer),采用Countstar或BD Rhapsody Scanner能够准确判断细胞数量以及活性状态。(注意:当红细胞/死细胞比例过多时,建议去除此类细胞,保证有效数据量。) BD Rhapsody Scanner检测图 注:活细胞检测明场图(左),绿色荧光(中),红色荧光(右) 3、单细胞捕获采用10X Genomics或BD Rhapsody单细胞捕获平台,将每个细胞及其mRNA标记上对应的标签。将收集的带上磁珠标签的RNA进行反转录,产物可于4℃环境下带回公司进行后续操作。 图一:Drop-Seq 哈佛医学院Steven McCarroll领导的团队将微流控技术引入单细胞RNA-seq方法中,开发了Drop-seq技术,且该项技术于2015年发表于《Cell》杂志上(Macosko et al., 2015)。Drop-seq技术利用微流体装置将带有细胞条形码的微珠和细胞一起装入微滴。这些液滴在一个小型设备上生成,沿着一根头发宽的槽道流动。条形码附着到每个细胞的一些基因上,因此科学家们可以一次测序所有的基因,追踪每个基因的来源细胞。 图二:10X Genomics 2016年,10X Genomics公司首次推出ChromiumTM系统,全面对接Illumina测序仪,能够自动化完成大规模单细胞研究。10X Genomics单细胞捕获平台起源自Drop-Seq技术,通过“双十字”微流控系统形成一个个含有细胞和凝胶微珠(gel bead)的油包水的乳滴(GEMs),其核心技术在于凝胶微珠表面的引物序列,由标记细胞的Barcode、标记细胞内mRNA的UMI和捕获mRNA的Poly dT组成。10X Genomics ChromiumTM系统可实现数千乃至数万个单细胞的分析,解决常规scRNA-seq在通量或扩展性方面存在的不足。 图三:BD Rhapsody BD Rhapsody™单细胞分析系统的诞生基于 BD 在细胞生物学领域 40年的专业技术, 采用CytoSeq特有的蜂窝板技术进行单细胞捕获。该技术用20W+的微孔(该数量级远大于Input细胞数量),保证单孔中的单细胞捕获。同时避免了传统微流控系统中存在的概率碰撞影响捕获效率的问题,采用微孔捕获相对会有更好的捕获效率,保证Input细胞的全面使用。 4、高通量测序PCR扩增后开始进行文库构建,获得每个样本的测序文库。接着采用Illumina Hiseq或NovaSeq高通量测序仪对单细胞转录组文库进行测序,获得每个样本的测序数据。 NovaSeq 6000测序仪 5、细胞数量判断采用Fastp软件对下机原始数据进行质控,对质控后测序数据中的cell barcode信息及其对应的counts数进行统计,判断测序样本中实际检测到的细胞数量,获得样本的测序细胞数,并根据最终确认的cell barcode信息提取对应的reads。 注:横坐标为细胞数量,纵坐标为每个细胞对应的平均counts数,根据曲线的斜率判断实际检测的细胞数量 6、基因组比对和表达量统计以cell barcode对应的reads为研究对象,采用STAR算法将测序数据比对到物种对应的基因组上,获得基因组比对的bam文件。再以bam文件以及基因组注释文件为研究对象,将比对到同一基因上的UMI进行合并,并去除其中重复的UMI序列,得到每个基因的UMI数量,统计每个细胞中检测到的基因数以及转录本数量,并得到表达量矩阵表。 注:左图为细胞中总共检测到的基因数量,右图为去除重复UMI后统计的基因数量 7、细胞过滤和数据标化利用基因组比对结果以及表达量结果对测序检测到的细胞进行过滤,去除细胞中基因检测数少、线粒体基因占比大的细胞,统计过滤后的细胞数量并得到对应的表达量矩阵表。采用数据标准化方法(CPM/RLE/UQ/TMM/scran/Downsampling等),对不同样本中细胞基因表达量进行标准化,得到标准化后的表达量矩阵表。 注:横坐标表示每个细胞中UMI的数量,纵坐标表示线粒体基因的占比情况 8、细胞亚群分析基于每个细胞中的基因表达量数据,采用聚类算法对细胞进行亚群分析,同时采用t-SNE分析对细胞的分群结果进行可视化展示。同时,还可以对不同样本中各细胞亚群的占比情况进行统计分析。 Azizi E et al., Cell. 2018 注:上图为乳腺癌肿瘤组织、正常组织、血液和淋巴结样本中免疫细胞亚群鉴定;下图为不同组织样本中各细胞亚群的占比情况 9、Marker基因鉴定鉴定不同细胞亚群中的Marker基因,并对Marker基因的表达分布进行可视化展示。 Dick S A et al., Nature immunology. 2019 注: marker基因的Feature Plot图(上)和Violin图(下) 10、差异基因筛选针对所有或者特定细胞亚群,进行细胞亚群间差异表达基因筛选,获得细胞亚群间差异表达基因。 Dick S A et al., Nature immunology. 2019 注:该图为不同细胞亚群间差异基因聚类分析图(Heatmap) 11、功能分析(GO Analysis)和信号通路分析(Pathway Analysis)分别采用NCBI/UNIPROT/SWISSPROT/AMIGO等GO数据库,以及KEGG数据库,对Marker基因/差异基因进行功能分析和信号通路分析,从而得到这些基因群体所显著性富集的GO条目和Pathway条目。 Dick S A et al., Nature immunology. 2019 注:该图为不同cluster之间差异基因显著富集的Pathway条目 1、RNA velocity分析采用velocyto算法,预测单个细胞的变化方向,得到细胞间的转变过程。 Manno G L et al., Nature, 2018 注:该图为RNA velocity分析结果图,图中箭头方向代表算法预测的细胞演化方向 2、Pesudotime分析以细胞的表达量数据为研究对象,采用TSCAN/monocle/SLICER/Ouija等算法,在虚拟时间轴上对细胞的变化模式进行分析,模拟重建细胞的动态变化过程,获得细胞间的状态转换关系,以及不同状态细胞间差异基因的表达情况。 Dick S A et al., Nature immunology. 2019 注:细胞间状态转换的pesudotime轨迹图(右)和Heatmap图(左) 3、细胞间通讯分析以细胞亚群的基因表达量数据为研究对象,获得细胞中的配体及受体基因的表达信息,得到细胞亚群间的信号通讯关系。 Vento-Tormo R et al., Nature. 2018 注:横坐标表示细胞类型,纵坐标表示细胞间通讯关系,圆圈大小表示显著性差异水平,圆圈颜色越红表示细胞间通讯关系越强 4、TCGA预后联合分析以TCGA临床信息以及筛选到的关键基因为研究对象,结合TCGA临床数据,进行预后分析,获得该基因/基因集与临床预后之间的关系。 Guo X, et al. Nature Medicine, 2018 注:横坐标表示生存期,纵坐标表示占比,不同颜色曲线代表不同分组 文献示例[1] Mickelsen LE, Bolisetty M, Chimileski BR, et al. Single-cell transcriptomic analysis of the lateral hypothalamic area reveals molecularly distinct populations of inhibitory and excitatory neurons[J]. Nat Neurosci. 2019 Apr;22(4):642-656.(IF=19.912) [2] Pijuan-Sala B, Griffiths JA, Guibentif C, et al. A single-cell molecular map of mouse gastrulation and early organogenesis[J]. Nature. 2019 Feb;566(7745):490-495.(IF=41.577) [3] Cao J, Spielmann M, Qiu X, et al. The single-cell transcriptional landscape of mammalian organogenesis[J]. Nature. 2019 Feb;566(7745):496-502.(IF=41.577) [4] Tiklová K, Björklund ÅK, Lahti L, et al. Single-cell RNA sequencing reveals midbrain dopamine neuron diversity emerging during mouse brain development[J]. Nat Commun. 2019 Feb 4;10(1):581.(IF=12.353) [5] Bartoschek M, Oskolkov N, Bocci M, et al. Spatially and functionally distinct subclasses of breast cancer-associated fibroblasts revealed by single cell RNA sequencing[J]. Nat Commun. 2018 Dec 4;9(1):5150.(IF=12.353) [6] Guo X, Zhang Y, Zheng L, et al. Global characterization of T cells in non-small-cell lung cancer by single-cell sequencing[J]. Nature Medicine. 2018 Jul;24(7):978-985.(IF=32.621) [7] Azizi E, Carr AJ, Plitas G, et al. Single-Cell Map of Diverse Immune Phenotypes in the Breast Tumor Microenvironment[J]. Cell. 2018 Aug 23;174(5):1293-1308.e36.(IF=31.398) [8] Fujii M, Matano M, Toshimitsu K,et al. Human Intestinal Organoids Maintain Self-Renewal Capacity and Cellular Diversity in Niche-Inspired Culture Condition[J]. Cell Stem Cell. 2018 Dec 6;23(6):787-793.e6.(IF=23.29) [9] Dick SA, Macklin JA, Nejat S, et al. Self-renewing resident cardiac macrophages limit adverse remodeling following myocardial infarction[J]. Nat Immunol. 2019 Jan;20(1):29-39.(IF=21.809) [10] Kumar M P, Du J, Lagoudas G, et al. Analysis of Single-Cell RNA-Seq Identifies Cell-Cell Communication Associated with Tumor Characteristics[J]. Cell Reports. 2018 Nov 6;25(6):1458-1468.e4.(IF=8.032) |