利用数据库数据进行定制化的生信分析
让您的文字更出彩
|
外显子测序外显子仅占人类基因组的2.5%,但包含了基因表达相关的绝大部分信息。该区域的变异与癌症、单基因遗传病、部分复杂疾病等密切相关。外显子捕获测序仅对外显子区域的序列进行测序,相对于基因组重测序成本较低,可以用很小的数据量获得大量有用信息,对研究基因的SNP、InDel等具有很大的优势。
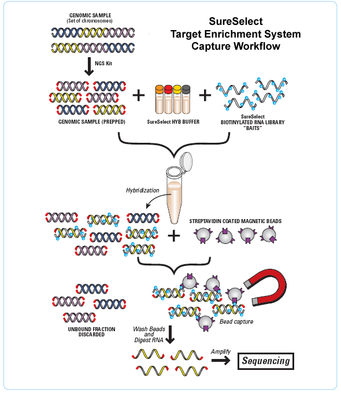
Aglient SureSelect外显子捕获系统 (www.Agilent.com) 我们的优势1. 整合已有数据库的同时,可以发现全新的突变位点; 2. 采用生物信息学领域权威的外显子数据分析流程,并有高分文献支持; 3. 整合最新的注释数据库,确保结果的准确性和高验证率; 4. 针对特定研究对象设计多套完善的备选方案,并推荐最优的一套。 样本要求组织样品 1. 新鲜动物组织干重≥0.5g 2. 新鲜植物组织干重≥1g 3. 新鲜培养细胞数≥8×106个 4. 全血(哺乳动物)≥2ml 5. 全血(非哺乳动物)≥0.5ml 6. 福尔马林固定石蜡包埋组织(FFPE)>10个玻片或50mm2,5-10μm厚的切片10片 DNA样品 1. 请提供总量≥3μg/样本,浓度≥50ng/μL的DNA; 2. OD260/280介于1.8-2.0之间; 3. 电泳检测无明显RNA污染,基因组条带清晰、完整,无降解; 4. 送样时请标记清楚样品编号,管口使用Parafilm膜密封; 5. 样品保存期间切忌反复冻融; 6. 送样时请使用干冰运输。 注:不同样品之间存在差异,详情请向我们咨询 实验流程
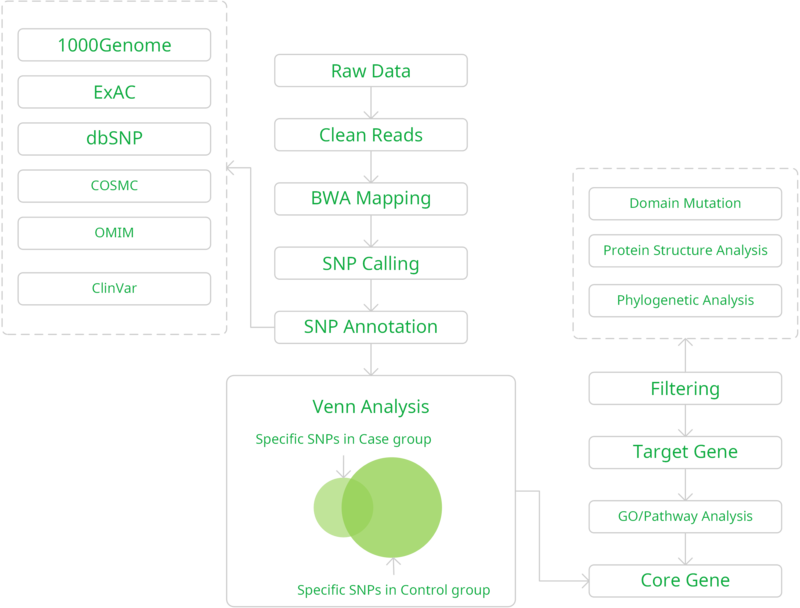
1. DNA提取及质控:凝胶电泳质控→Nanodrop质控→Agilent 2200质控; 2. 靶向捕获:Agilent Sureselect V6外显子捕获,90分钟内完成杂交; 3. 引物序列降解:去除引物序列的影响; 4. 文库构建及质控:PCR扩增; 5. 上机测序:建议选择NovaSeq,双端测序,通量大,碱基精度高,而且成本低,速度快。测序数据量:10Gb。 数据分析流程
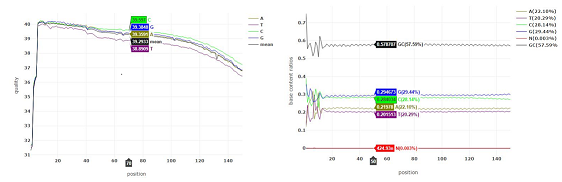
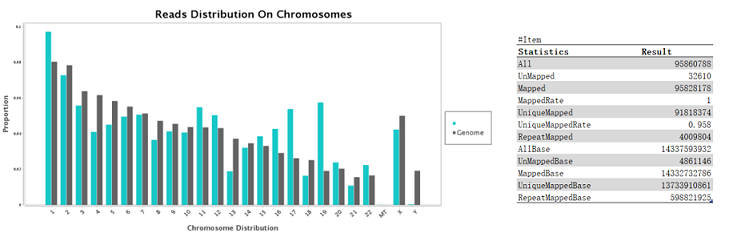
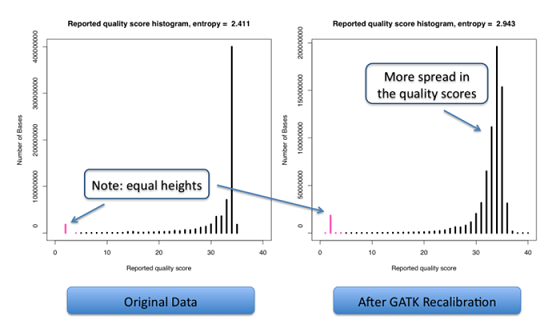
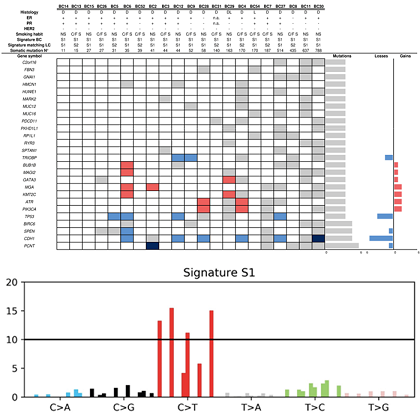
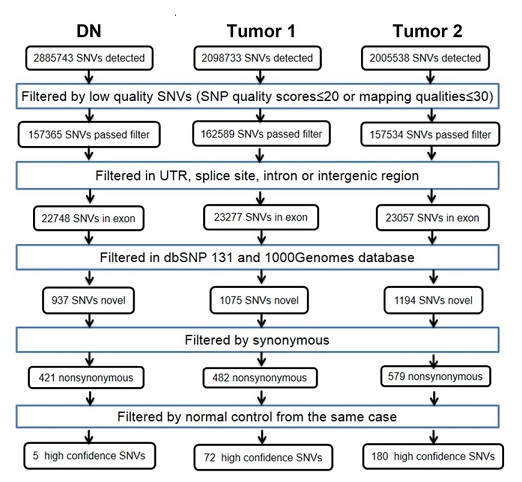
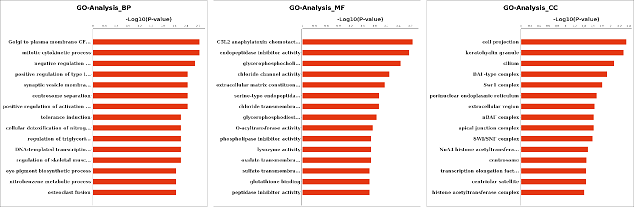
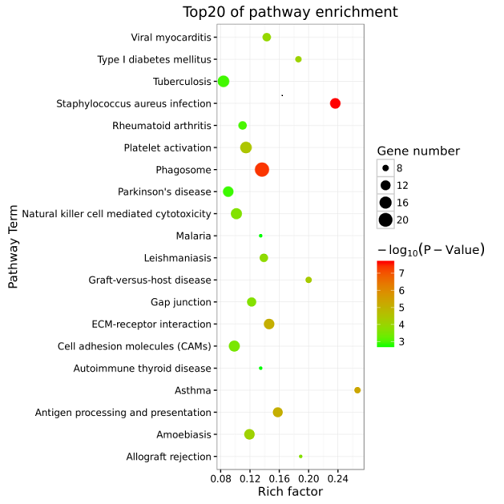
结果示例1、原始数据质量控制以原始数据为研究对象,采用Fastp软件对于低质量序列,未检测序列,接头序列进行过滤,并对于过滤前后数据的GC比值,碱基质量,长度分布,接头留存,Duplication比率等指数进行分析。 原始数据质量控制结果 注:左图横坐标代表碱基位点,纵坐标代表碱基质量值,不同颜色曲线代表不同碱基在每条read上的质量值;右图横坐标代表碱基位点,纵坐标代表碱基含量比值,不同颜色曲线代表不同位点各碱基含量。 2、DNA基因组比对(DNA Mapping)与质控采用BWA-mem/Bowtie2等算法将质控后的测序数据比对到基因组上,得到基因组比对的bam文件。并基于bam文件进行信息统计,得到reads在染色体上的分布、基因组比对率信息。 DNA Mapping结果 注:左图为reads在染色体上的分布情况;右图为reads与参考基因组序列比对情况 3、DNA基因组比对优化采用Samtools和GATK tools,对基因组比对的bam文件进行Remove Duplicate和Recalibrate分析,得到每一个样本优化后的Bam文件。 基因组比对结构优化 注:该图展示了GATK tools矫正前后的reads碱基质量值 4、体细胞突变分析(SNP Calling)采用Mutect2等算法,进行突变分析(SNP Calling),得到肿瘤相对于癌旁的体细胞突变结果。 SNP Calling Jiang et al., Hepatology, 2014 注:IGV确认候选体细胞突变位点图 5、体细胞突变注释采用VEP软件对突变发生位点所在的基因进行注释,并对于突变类型以及突变位点在多种数据库中的记载情况进行展示。 体细胞突变基因注释及突变类型 Coco S et al., Cancers, 2019 注:上图为肿瘤组织样本中的突变基因;下图为突变类型的突变频率,横轴为突变类型,纵轴为特定突变类型的突变频率 6、突变过滤采用行业标准的肿瘤体细胞突变过滤策略,对所有的SNP注释进行突变过滤,得到具有研究意义的SNP位点以及其注释信息。 SNP/SNV筛选 Jiang et al., Hepatology, 2014 注:该图展示了肿瘤体细胞SNP/SNV突变的过滤策略 7、突变基因功能分析(GO Analysis)采用NCBI/UNIPROT/SWISSPROT/AMIGO等GO数据库,对于突变基因进行功能分析得到突变基因所显著性富集的功能条目。 所有差异基因的GO分析结果 注:该图展示了显著性富集的前15个GO条目,横坐标表示-log10(P-value),纵坐标表示GO条目名称。 8、突变基因信号通路分析(Pathway Analysis)采用KEGG数据库,对于突变基因进行信号通路分析得到突变基因所显著性富集的信号通路条目。 Pathway富集性散点图 注:该图展示了显著性富集的20条Pathway条目。横坐标表示pathway对应的Rich factor,纵坐标表示pathway注释信息,点的颜色表示P-value的大小,点的大小表示pathway包含的差异基因数量。 1、构建系统发育树选取多个物种的同源蛋白,构建系统发育树,进一步直观了解该蛋白基因在不同物种中的保守情况及进化史。
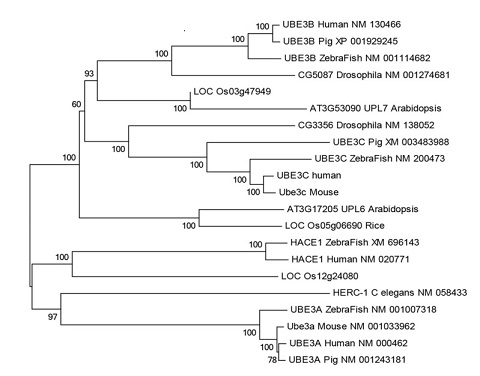
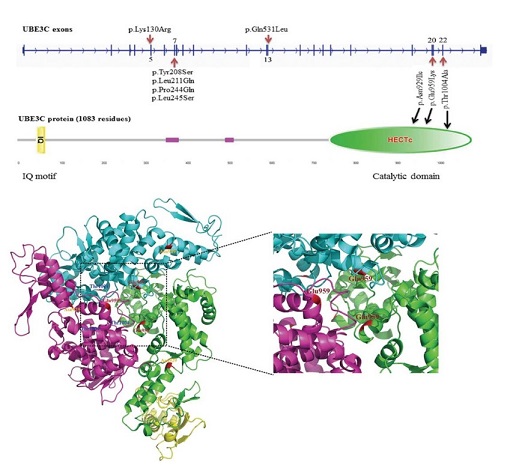
系统发育树 Jiang et al., Hepatology, 2014 注:该系统发育树表示含有HECT结构域的旁系同源物。scale bar表示每个位点氨基酸变化数,每个节点处的数值表示相应的bootstrap value。 2、构建蛋白质模型在蛋白活性中心或者修饰位点发生的突变往往会极大的影响蛋白的功能,因此通过蛋白质的3D模式图可找到位于蛋白活性中心的突变。 蛋白质模型 Jiang et al., Hepatology, 2014 注:采用生物信息学手段模拟蛋白质的结构并建立了模型,分析显示Glu959突变位于HECT domain表面的α螺旋上,可能和蛋白活性位点有关。 3、进化分析将目的基因与其他物种以及本物种的同源基因进行氨基酸保守性预测,观察发生突变的位点是否为保守的氨基酸位点,如果保守,则该位点的突变可能会影响蛋白功能。 进化分析 Jiang et al., Hepatology, 2014 注:该图展示了来自不同物种的UBE3Cparalogs的多重序列比对结果。其中,UBE3C中的突变位点用红色箭头标记。 文献示例[1] Xu LX, He MH, Dai ZH, et al. Genomic and transcriptional heterogeneity of multifocal hepatocellular carcinoma. Ann Oncol. 2019 Mar 27. pii: mdz103. (IF=13.926) [2] Ricciuti B, Kravets S, Dahlberg SE, et al. Use of targeted next generation sequencing to characterize tumor mutational burden and efficacy of immune checkpoint inhibition in small cell lung cancer. J Immunother Cancer. 2019. (IF=8.374) [3] Coco S, Bonfiglio S, Cittaro D, et al. Integrated Somatic and Germline Whole-Exome Sequencing Analysis in Women with Lung Cancer after a Previous Breast Cancer. Cancers (Basel). 2019 Mar 28;11(4). pii: E441. (IF=5.326) [4] Cho SY, Chae J, Na D, et al. Unstable Genome and Transcriptome Dynamics during Tumor Metastasis Contribute to Therapeutic Heterogeneity in Colorectal Cancers. Clin Cancer Res. 2019 Jan 22. (IF=10.199) [5] Jiang J, et al. Clinical Significance of the Ubiquitin Ligase UBE3C in Hepatocellular Carcinoma Revealed by Exome Sequencing. Hepatology. 2014 Jun;59(6):2216-27. (IF=14.079) |