Small RNA主要包括miRNA、piRNA、tsRNA(tRF&tiRNA)、snRNA和snoRNA等,是一类不具有蛋白编码能力的RNA分子,能调控基因表达,在细胞生长、发育和代谢等基础生物学过程中都扮演着重要的角色,甚至在癌症等相关疾病形成过程中也起着关键的作用。高通量测序技术省去了烦琐的Small RNA克隆文库构建过程,可以一次性生成上百万条Small RNA序列,能够快速鉴定特定条件下表达的已知Small RNA并发现新的Small RNA,同时还可以研究不同条件下Small RNA的表达差异,并可以联合转录组测序表达谱数据进行关联分析。
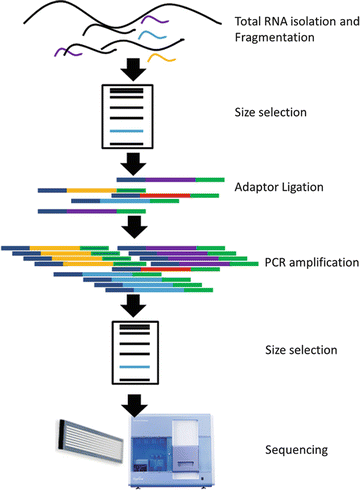
A workflow for Small RNA Sequencing
Jai PM et al., RNA Mapping, 2014
我们的优势
1. 针对动物(包括外泌体)Small RNA(miRNA、piRNA、tsRNA)提供定制化实验分析方案;
2. 不仅能高效准确发现并注释非模式物种新的miRNA,还能进行piRNA、tiRNA、tRF等最新的研究;
3. 及时更新miRBase、miRanda、RNAhybrid等专业miRNA数据库以及靶基因预测算法,保证分析结果紧跟行业前沿。
样本要求
组织样品:
1. 动物组织:>200mg;
2. 植物组织:>200mg;
3. 细胞培养:>2x106个;
4. 全血:>3ml;
5. 菌体:>2x106个或>300mg。
RNA样品:
1. 请提供体积15 μL - 100 μL,总量≥10μg,浓度≥ 200ng/μL的RNA;
2. OD260/280介于1.8~2.2之间,OD260/230≥2.0,RIN≥6.5,28S:18S≥1.0,确保RNA无降解;
3. 送样时请标记清楚样品编号,管口使用Parafilm膜密封;
4. 样品保存期间切忌反复冻融;
5. 送样时请使用干冰运输。
不同样品之间存在差异,详情请向我们咨询
实验流程





1. 总RNA提取及质控:RNA总量 > 1 μg ;RIN> 6.0
2. 小RNA富集:切胶范围10-50bp
3. 小RNA质控:Agilent 2200质控
4. 文库构建:文库分子18-30bp
5. 上机测序:Small RNA测序NovaSeq、HiSeq、Ion Proton等测序仪都可以完成。测序数据量40M reads。
数据分析流程
>>动物小RNA
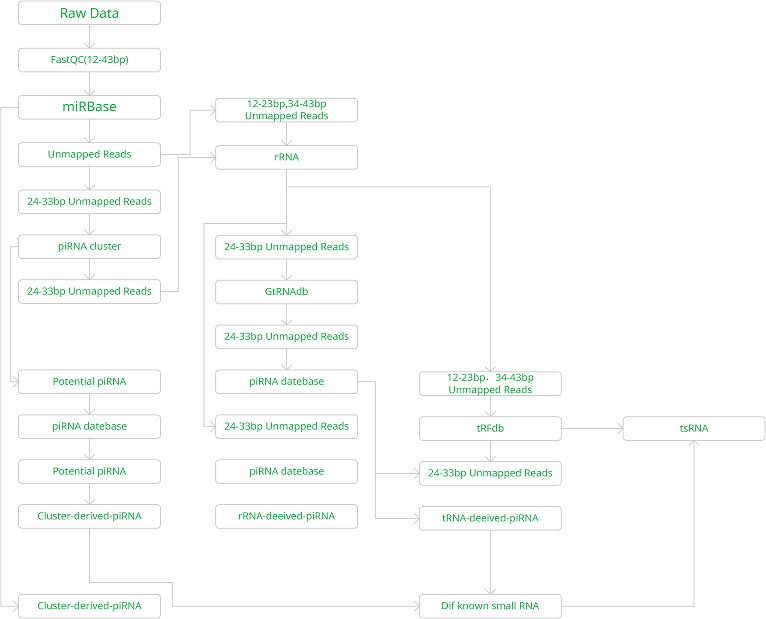
>>植物小RNA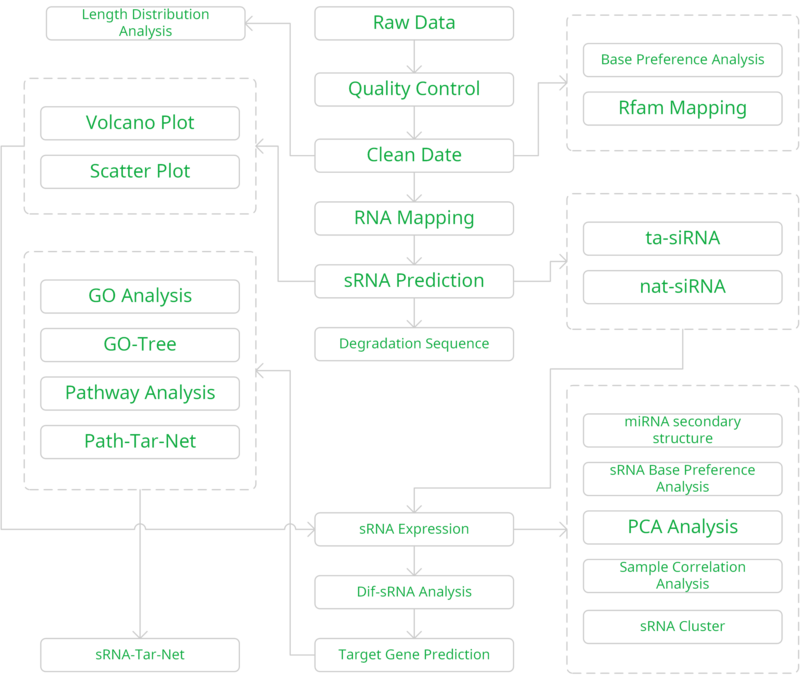
1、数据过滤对于原始数据进行质量控制,排除序列中的过短序列,低质量序列,未测出序列,并去掉双端接头序列,以保证测序数据的质量与准确性,我们将从碱基质量,GC含量等角度对测序数据进行质量评估,过滤后的数据称为Clean Data(过滤后数据)。
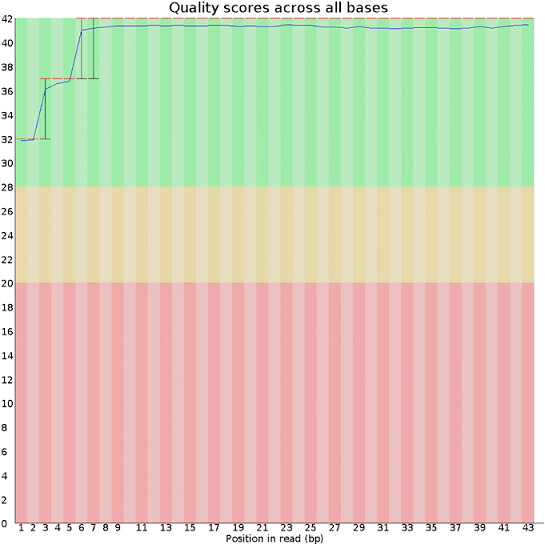
碱基质量结果图(过滤后)
注:横坐标表示碱基位点,纵坐标表示碱基质量值。
2、序列比对和长度分析由于Small RNA中成员较多,且各成员有各自的长度区间,我们首先会将测序得到的数据比对到分析物种的miRNA数据库中,再将unmapped reads比对到NCBI数据库中,对比对到不同数据库中的small RNA(miRNA、piRNA和tsRNA)序列进行长度分布统计,观察各类small RNA的长度分布情况以及主峰所在的位置。

small RNA长度分布情况
注:左图为miRNA长度分布图;右图为piRNA长度分布图。
3、碱基偏好性分析成熟的miRNA在首位往往就具有U的偏好性,同时来源于生殖细胞的piRNA,其首位也通常具有U偏好性。因此,在进行序列比对后,对于比对到不同数据库中的small RNA序列进行长度分布统计,观察各类small RNA的碱基偏好情况。
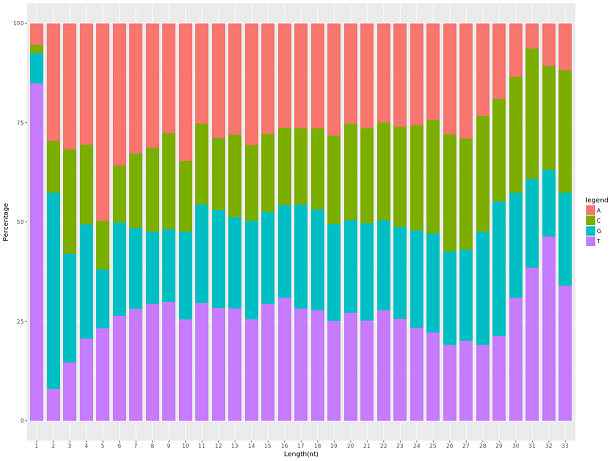
small RNA碱基偏好性分析
注:横坐标表示序列长度,纵坐标表示百分比,不同颜色表示不同的核苷酸。
4、表达量统计Small RNA测序最关键的步骤是对small RNA进行定量表达和差异筛选,将测序数据比对到各small RNA对应的数据库后,采用表达量统计软件对于每一个small RNA的Counts数进行标化。

miRNAs表达量统计分析
An X et al., Theriogenology, 2016
注:该表格列举了部分差异miRNAs的名称和序列信息,NE(normalized expression)表示标化后的miRNA表达量。
5、差异Small RNA筛选找到差异small RNA是整个研究的基础,常用的差异筛选算法有DESeq、edgeR等,将表达量统计的结果进行差异筛选,计算实验组 VS 对照组的差异情况,获得差异倍数、显著性(P-Value)以及误判率(FDR),通过这些指标筛选出显著性差异的Small RNA。
差异miRNAs热图
An J et al., Investigative Ophthalmology & Visual Science, 2015
注:横轴表示组别,纵轴表示不同miRNAs。
6、靶基因功能分析(GO Analysis)和信号通路分析(Pathway Analysis)采用NCBI/UNIPROT/SWISSPROT/AMIGO等GO数据库,以及KEGG数据,对靶基因进行功能和信号通路分析,得到靶基因所显著性富集的功能条目和Pathway条目,并绘制集靶基因、功能和pathway于一体的network。
miRNA靶基因功能和pathway网络图
An J et al., Investigative Ophthalmology & Visual Science, 2015
注:该图表示miRNA-204靶向的基因,及其靶基因所显著富集的GO条目(右)和Pathway条目(左)。
1、Novel miRNA预测非模式物种中存在很多未被报道的Novel miRNA,将其找出并进行研究是一项重要内容,NovelBio团队协助研究者将比对到基因组的reads提取出来,然后用行业内公认的算法,通过茎环结构自由能的计算,以及其他物种miRBase数据库的比对,最后获得预测的Novel miRNA。
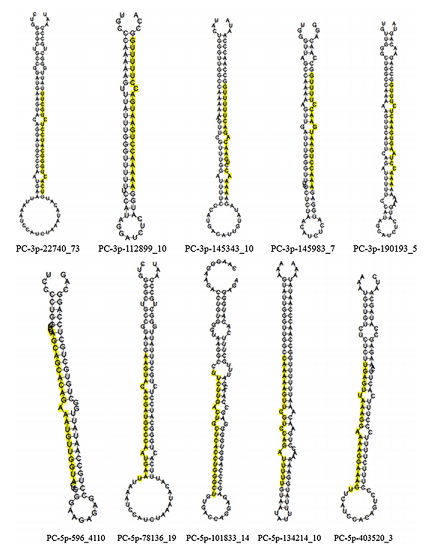
novel miRNAs的茎环结构
An X et al, Theriogenology. 2016
注:该图展示了进一步分析与已知miRNA不匹配的测序reads预测的新的miRNA,这10个序列都具有显著的茎-环发夹二级结构。
2、Small RNA靶基因预测以差异的small RNA为研究对象,采用miranda算法与RNAHybrid算法进行靶基因预测,得到small RNA靶向到mRNA的3’UTR序列的靶基因结合位点,并绘制靶基因网络图。
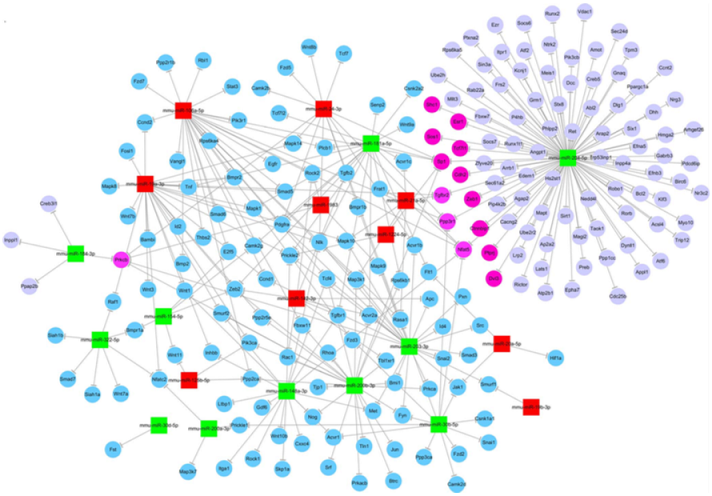
miRNAs靶基因网络图
An J et al., Investigative Ophthalmology & Visual Science, 2015
注: 绿色表示下调的miRNAs,红色表示上调的miRNAs。
3、Small RNA趋势分析以各分组间的miRNA的TPM.txt文件为研究对象,采用STEM算法,进行趋势分析,得到按照样本逻辑顺序所在趋势
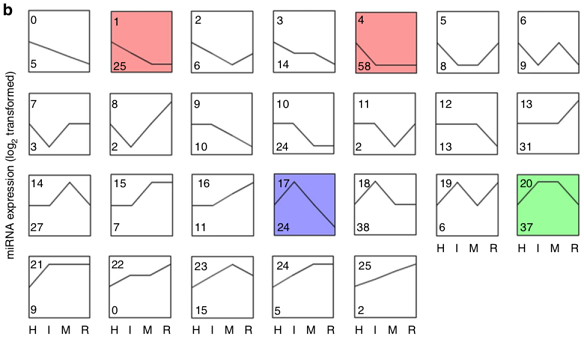
DCs分化过程中miRNAs表达情况
Su XP et al., Nature Communications, 2013
注:该图展示了DCs分化过程中miRNAs表达情况。H、I、M和R分别代表分化的四个阶段:H—HSCs;I—imDCs;M—maDCs;R—DCreg,左上角的数字代表将391个miRNAs分成了26个clusters,左下角的数字代表每个cluster中miRNAs的数量,有颜色的clusters代表miRNAs显著富集。
文献示例
[1] Zheng L, Zhang X, Zhang H, et al. The miR164-dependent regulatory pathway in developing maize seed. Mol Genet Genomics. 2019 Jan 3. (IF=2.734)
[2] Xu C, Zhang H, Zhou W, et al. MicroRNA-10a, -210, and -563 as circulating biomarkers for ossification of the posterior longitudinal ligament. Spine J. 2018 Oct 20. pii: S1529-9430(18)31165-3. (IF=3.119)
[3] Wang Y, Zhang CY, Xia RH, et al. The MYB/miR-130a/NDRG2 axis modulates tumor proliferation and metastatic potential in salivary adenoid cystic carcinoma. Cell Death Dis. 2018 Sep 11;9(9):917. (IF=5.638)
[4] Chen X, Hao Z, Wei G, et al. The microRNA-10a/ID3/RUNX2 axis modulates the development of Ossification of Posterior Longitudinal Ligament. Sci Rep. 2018 Jun 15;8(1):9225. (IF=4.122)
[5] He Q, et al. Downregulation of miR-7116-5p in microglia by MPP1 sensitizes TNF-a production to induce dopaminergic neuron damage. Glia. 2017 Aug;65(8):1251-1263. (IF=6.2)
[6] Xu C, et al. Integrated microRNA-mRNA analyses reveal OPLL specific microRNA regulatory network using high-throughput sequencing. Sci Rep. 2016 Feb 12;6:21580. (IF=5.578)
[7] Liu R, et al. DNMT1-microRNA126 epigenetic circuit contributes to esophageal squamous cell carcinoma growth via ADAM9-EGFR-AKT signaling. Clin Cancer Res. 2015 Feb 15;21(4):854-63. (IF=8.193)
[8] Cheng T, et al. MeCP2 Suppresses Nuclear MicroRNA Processing and Dendritic Growth by Regulating the DGCR8/Drosha Complex. Dev Cell. 2014 Mar 10;28(5):547-60. (IF=14.08)
[9] Su X, et al. miRNomes of haematopoietic stem cells and dendritic cells identify miR-30b as a regulator of Notch1. Nat Commun. 2013;4:2903. (IF=10.02)
[10] Xu C, et al. miRNA -100 Inhibits Human Bladder Urothelial Carcinogenesis by Directly Targeting mTOR. Molecular Cancer Therapeutics. 2013, Feb 12:207-219. (IF=5.599)
